在前面文章中介绍了独立样本t检验(Independent Samples t-test)的假设检验理论 ,本篇文章将使用实例演示在R软件中实现独立样本t检验的操作步骤。
关键词:R语言; R软件; t检验; 独立样本t检验; 成组t检验; 两样本均数比较; 近似t检验; 韦尔奇t检验; Welch近似t检验
一、案例介绍
某医生研究某生化指标(X)对病毒性肝炎诊断的临床意义,测得20名正常人和19名病毒性肝炎患者生化指标(X)含量(μg/dl),问病毒性肝炎患者和正常人生化指标(X)含量是否存在差异?部分数据见图1。本文案例可从“附件下载”处下载。
二、问题分析
本案例的分析目的是比较两组数据均值是否有差异,即判断病毒性肝炎患者和正常人生化指标(X)含量是否存在差异。针对这种情况可以使用独立样本t检验。但需要满足6个条件:
条件1:观察变量为连续变量。本研究中的生化指标含量为连续变量,该条件满足。
条件2:观察变量相互独立。本研究中各研究对象的观察变量都是独立的,不存在互相干扰的情况,该条件满足。
条件3:观察变量可分为2组。本研究中分为病毒性肝炎患者和正常人,该条件满足。
条件4:观察变量不存在显著的异常值,该条件需要通过软件分析后判断。
条件5:各组观察变量为正态(或近似正态)分布,该条件需要通过软件分析后判断。
条件6:两组观察变量的方差相等,该条件需要通过软件分析后判断。
三、软件操作及结果解读
(一) 导入数据
mydata <- read.csv("独立样本t检验.csv") #导入CSV数据
View(mydata) #查看数据
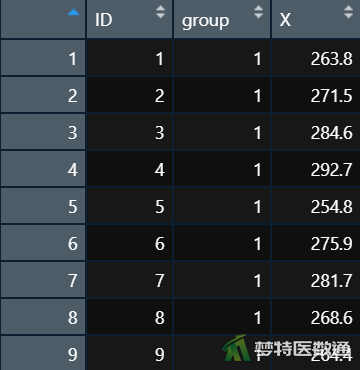
在数据栏目中可以查看全部数据情况,数据集中共有3个变量和39个观察数据,3个变量分别代表被调查者的编号(ID)、分组(group)及其对应的生化指标(X)。
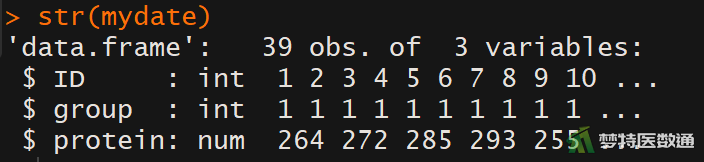
如果数据集较大也可使用如下命令查看数据框结构:str(mydata) #查看数据框结构
(二) 适用条件判断
1.条件4判断(异常值判断)
(1) 软件操作
##拆分数据##
mydata1<-subset(mydata, mydata$group==1) #按组生成数据子集1 mydata2<-subset(mydata, mydata$group==2) #按组生成数据子集2
## 分组描述数据子集 ##
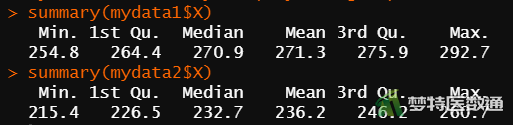
summary(mydata1$X) #描述子集1基本情况 summary(mydata2$X) #描述子集2基本情况
##分组查看缺失值情况##
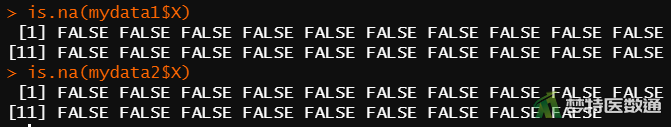
is.na(mydata1$X) #查看子集1有无缺失值 is.na(mydata2$X) #查看子集2有无缺失值
##分组绘制箱线图##
labels <- c("Normal","Hepatitis") #设置分组标签
boxplot(mydata$X ~ mydata$group, names = labels,
xlab = c("group"), ylab = expression("X")) #分组绘制箱线图
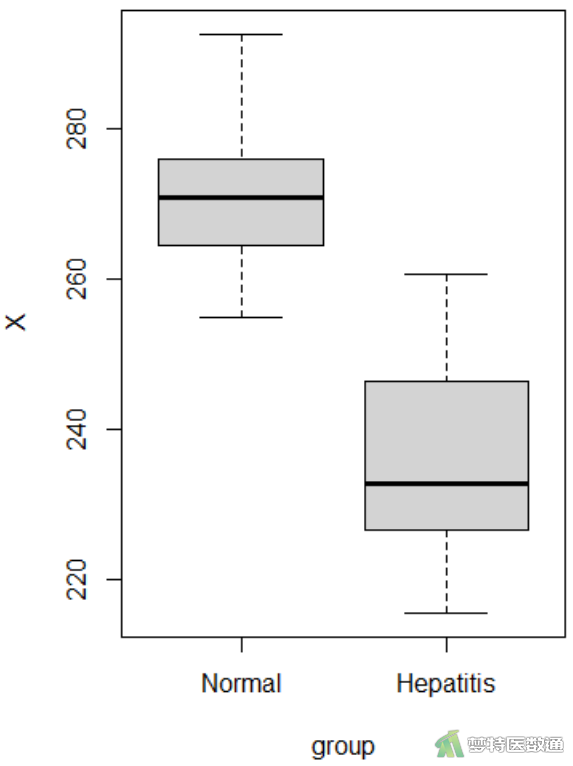
(2) 结果解读
图3“summary (描述性分析)”命令运行结果,列出了观察变量的“Min(最小值)”、“1st Qu(P25)”、“Median(中位数)”、“Mean(平均值)”、“3rd Qu(P75)”和“Max(最大值)”,依据专业可判断人体生化指标含量均可能存在215.4μg/dl和292.7μg/dl的情况;此外,图5中的箱线图也未提示任何异常值。综上,本案例未发现需要删除的异常值,满足条件4。
2. 条件5判断(正态性检验)
(1) 软件操作
par(mfrow = c(1, 2)) #设置画1行2个图片 qqnorm(mydata1$X, ylab="X", main="Normal") #绘制子集1的qq图 qqline(mydata1$X) #增加趋势线 qqnorm(mydata2$X, ylab="X", main="Hepatitis") #绘制子集2的qq图 qqline(mydata2$X) #增加趋势线
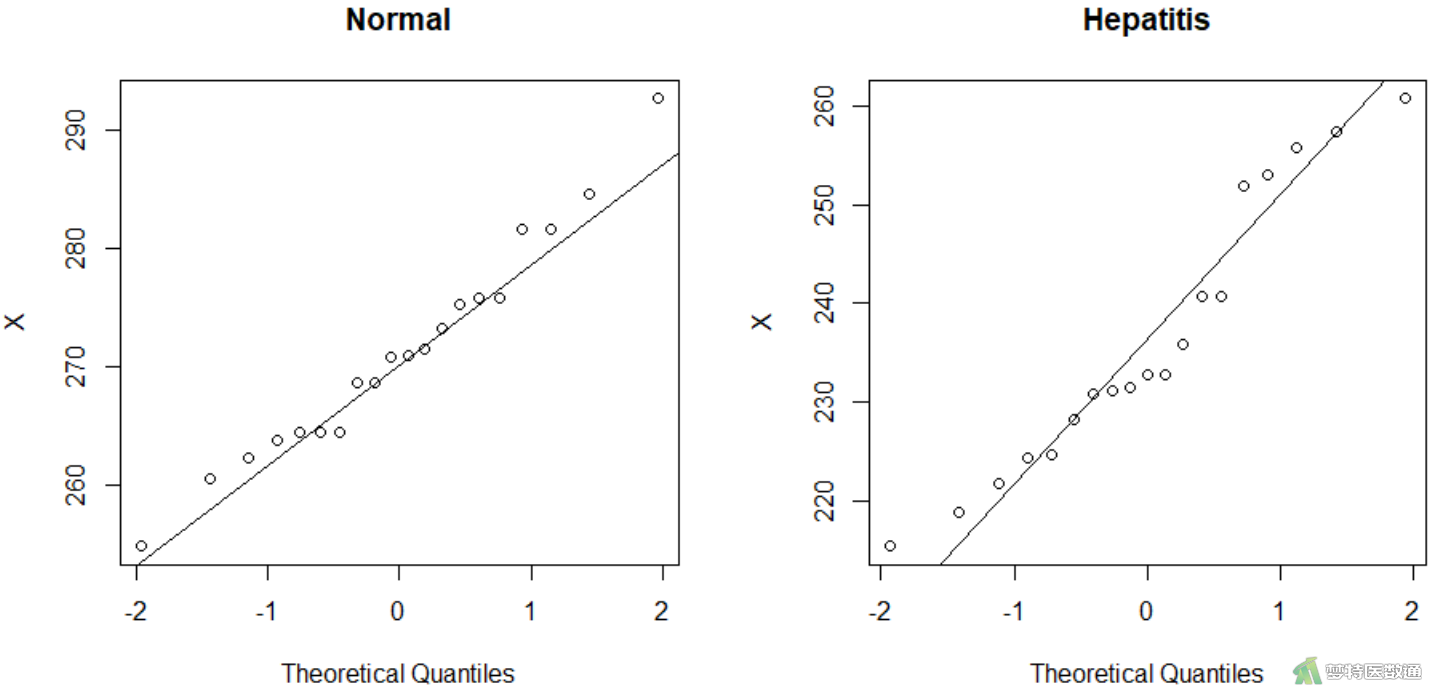
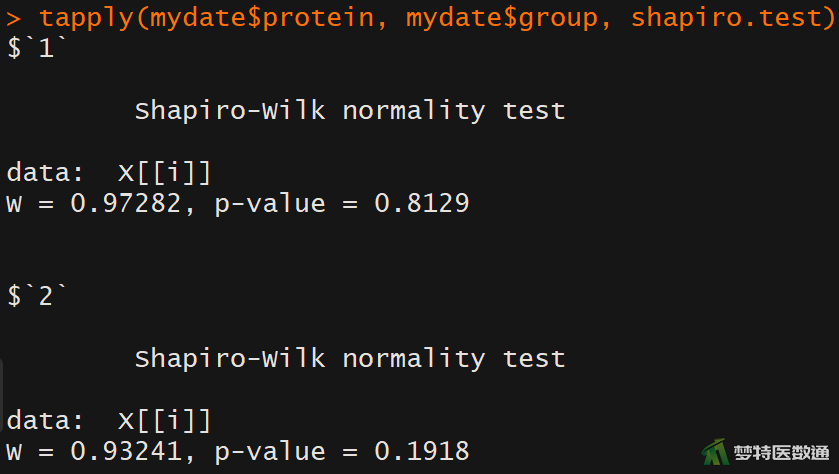
tapply(mydata$X, mydata$group, shapiro.test) #分组进行Shapiro-Wilk正态性检验
(2) 结果解读
图6 Q-Q图上两组散点基本围绕对角线分布,提示两组数据均呈正态分布;图7的“Shapiro-Wilk normality test (S-W正态性检验)”表格结果显示P=0.8129和0.1918,均>0.1,也提示两组数据均服从正态分布。综上,本案例满足条件5。关于正态性检验的更多内容请阅读医学统计学核心概念及重要假设检验的软件实现(2/4) ——正态性假设检验的SPSS实现。
3. 条件6判断(方差齐性检验)
(1) 软件操作
##计算标准差##
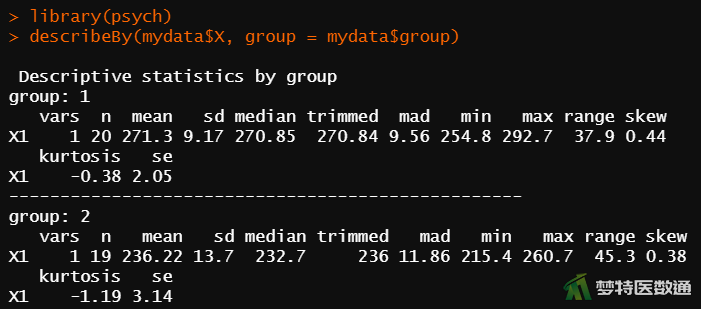
library(psych) #调用包“psych” describeBy(mydata$X, group = mydata$group) #分组描述统计
##levene法方差齐性检验##
library(dplyr) #调用软件包“dplyr”
mydata3 <- mydata %>% mutate(group1=factor(group,labels=c("Normal","Hepatitis"))) #把变量group转换为分类变量
library(car) #调用软件包“car”
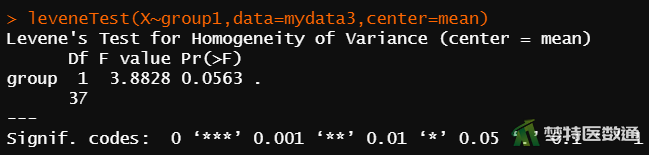
leveneTest(X~group1,data=mydata3,center=mean) #levene法方差齐性检验
(2) 结果解读
图8“describe (描述性分析)”运行结果提供了各组观察变量的统计量,包括:“vars(第几个变量)”“n (样本量)”、“mean (均数)”、 “sd (标准差)”、“median (中位数)”、“terimmed(截尾均值)”、“mad (绝对中位差)”、“min(最小值)”、“max(最大值)”、range(全距)、“skew(偏度)”、“kurtosis(峰度)”和“se (标准误)”。 结果可知,正常人组和肝炎患者组的标准差分别为9.17和13.70,两组之间标准差直观看来似乎存在差异,但还需要依据统计学检验的结果进行综合判断。图9 “LeveneTest(Levene检验)”结果显示,F=3.8828,P=0.0563<0.1,提示两组数据的方差不齐,不满足条件6。关于方差齐性检验的更多内容请阅读医学统计学核心概念及重要假设检验的软件实现(4/4)——方差齐性检验及SPSS实现。
(三) 统计描述及推断
1.软件操作
install.packages(“ggpubr”) #安装包“ggpubr”
library(ggpubr) #调用包“ggpubr”
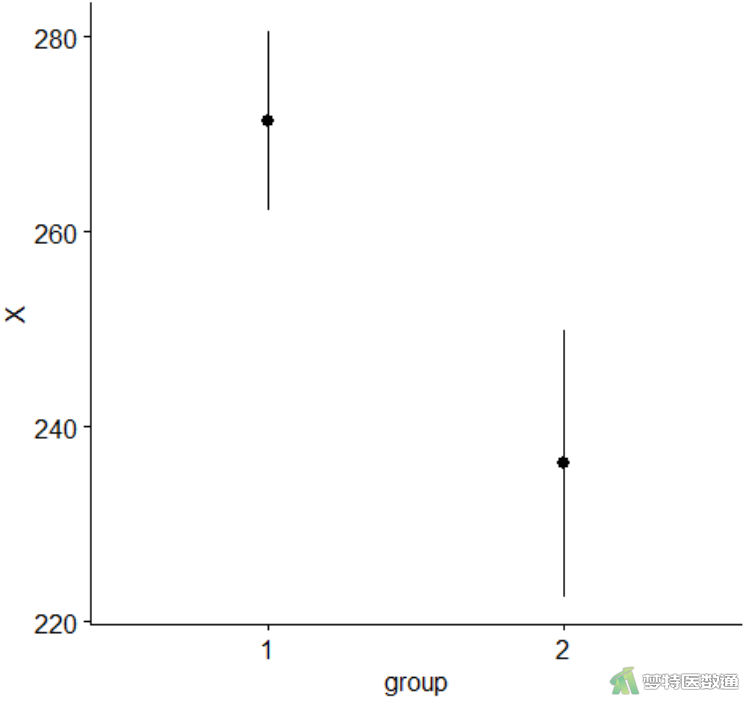
ggerrorplot(mydata, x = "group", y = "X",
desc_stat = "mean_sd") #绘制平均值标准差图
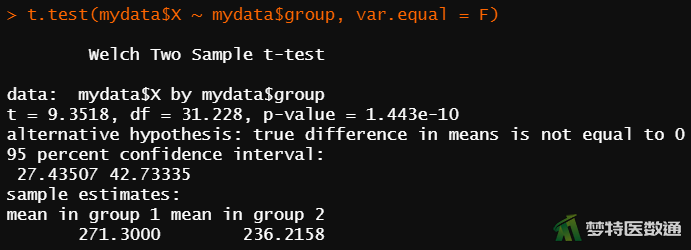
t.test(mydata$X ~ mydata$group, var.equal = F) #独立样本t检验
2. 结果解读
(1) 统计描述
图8“DescribeBy (描述性分析)”的部分运行结果提供了研究案例的“Group (组别)”、“N (样本量)”、“Mean (均数)”、“Median (中位数)”、“SD (标准差)”和“SE (标准误)”。可知,正常人群的生化指标含量为271.30±9.17μg/dl,肝炎患者的生化指标含量为236.22±13.70μg/dl。图10为两组人群生化指标含量的分布图。两组的生化指标含量貌似存在差异,但还需要依据统计学检验的结果进行判断。
(2) 统计学推断
由于本案例满足正态性要求,但不满足方差齐性要求,所以采用校正t检验(Welch Two Sample t-test)分析结果。可知肝炎患者的生化指标含量平均值比正常人群低35.084μg/dl,95%CI为27.435~42.733;差异有统计学意义(t’=9.3518,P<0.001)。
四、结论
本研究采用独立样本t检验判断病毒性肝炎患者和正常人生化指标含量是否存在差异。通过专业知识判断,两组数据不存在需要删除的异常值;通过绘制Q-Q图和Shapiro-Wilk检验,提示两组数据服从正态分布;通过Levene检验,提示两组数据间方差不齐,采用Welch’s t检验对数据进行分析。
结果显示,正常人群和肝炎患者的生化指标含量分别为271.30±9.17μg/dl和236.22±13.70μg/dl;肝炎患者的生化指标含量平均值比正常人群低35.084μg/dl (95%CI:27.435~42.733),差异有统计学意义(t’=9.3518,P<0.001)。因此,可以认为该生化指标含量对病毒性肝炎的临床诊断具有价值。