在前面文章中介绍了配对样本Wilcoxon符号秩检验(Paired Samples Wilcoxon Signed Rank Test)的假设检验理论,本篇文章将实例演示在SAS软件中实现配对样本Wilcoxon符号秩检验的操作步骤。
关键词:SAS; 非参数检验; 秩和检验; 配对样本Wilcoxon符号秩检验; 配对秩和检验
一、案例介绍
对12份血清分别用原方法(检测时间15分钟)和新方法(检测时间10分钟)测谷草转氨酶,问两种方法所得结果是否有差别?数据见图1。本文案例可从“附件下载”处下载。
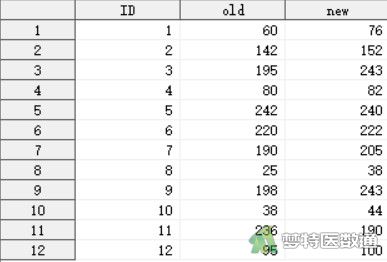
二、问题分析
本案例的分析目的是比较对同一组样品使用两种方法检测的结果是否有差异,即判断用新法与原法检测血清谷草转氨酶含量是否存在差异,属于配对设计定量资料比较的范畴。对于配对设计的连续性变量比较,可以选用配对t检验或Wilcoxon符号秩检验。配对t检验适用于两组差值近似服从正态分布的数据。当不满足该条件时,可选择Wilcoxon符号秩检验。首先,对新法与原法检测血清谷草转氨酶含量的差值进行正态性检验,若发现差值不服从正态分布,则应选用配对样本Wilcoxon符号秩检验。使用Wilcoxon 符号秩检验时,需要满足3个条件:
条件1:观察变量是连续变量或有序分类变量。本研究中的谷草转氨酶水平为连续变量,该条件满足。
条件2:观察变量可分为2组。本研究中分为原法和新法,该条件满足。
条件3:观察变量的数据结构为配对形式。本研究中数据属于同一组样品自身配对的形式,该条件满足。
三、软件操作及结果解读
(一) 导入数据
①利用LIBNAME语句建立SAS逻辑库关联,注意逻辑库名称要求,即最大长度8字符,必须以字母或下划线“_”开始,可以是字母、数字和下划线的任意组合。具体代码如下:
libname mydata 'D:\mydata';
通过这一步骤,SAS能够识别引号中的物理位置,将逻辑库建立在该目录下,同时在以下过程中新建的SAS表格便可以永久储存在该位置,便于反复读取和使用。先运行该代码使其生效。
②利用PROC IMPORT语句导入文件,代码如下:
proc import out= mydata.example datafile=" D:\mydata\配对样本Wilcoxon符号秩检验.csv" dbms=csv replace; getnames=yes; run;
该过程在mydata逻辑库中生成example数据集,数据文件由DATAFILE=选项指定,DBMS=选项指定其数据库类型。该案例中初始数据集为csv文件,故而使用“dbms=csv”指定。如果已经存在相同名称的SAS数据集,即可使用REPLACE选项进行覆盖。GERNAMES=YES选项指定从第2行开始读取数据,将数据集的首行变量名作为SAS数据集的变量名。
(二) 适用条件判断(正态性检验)
1. 软件操作
配对样本Wilcoxon符号秩检验时,需要考察配对组别观测变量差值的正态性情况。所以正态性检验前先计算配对组别观测变量的差值(即new-old),将差值命名为change,数据见图2。代码如下:
data mydata.example; set mydata.example; change=new-old; run;
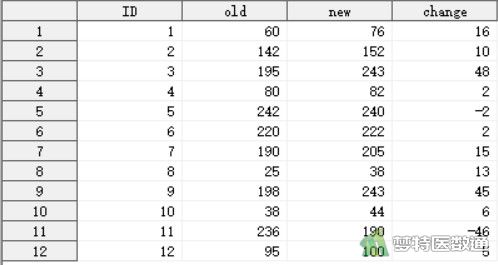
接下来对表示两组差值的变量“Change”进行正态性检验,代码如下:
proc univariate data= mydata.example plot normal; var change; Histogram /normal kernel; run;
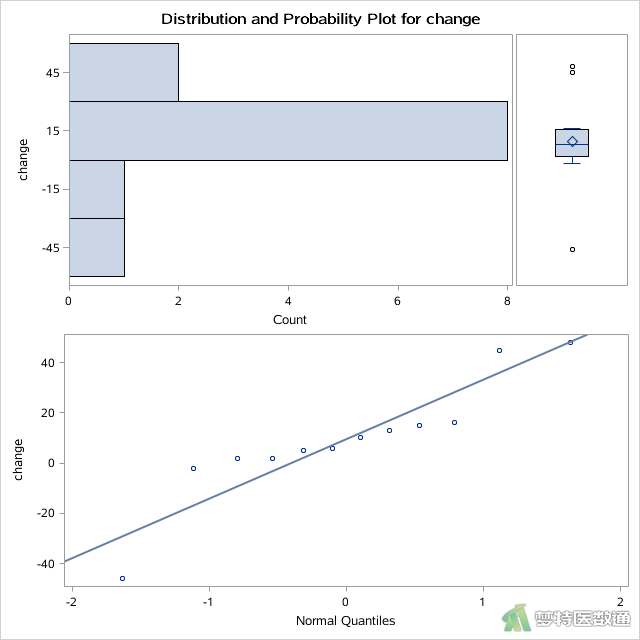
其中“mydata.example”为上一步中导入的数据集名称,“change”为分析变量名,即配对组别观测变量的差值。选项PLOT产生了平行条状图,箱线图和正态概率图,如图3所示。
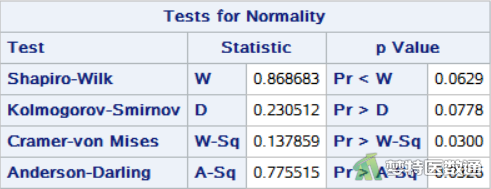
选项NORMAL进行正态性检验,得到“Normality Test (Shapiro-Wilk) (夏皮罗-威尔克正态性)”检验结果,如图4所示。
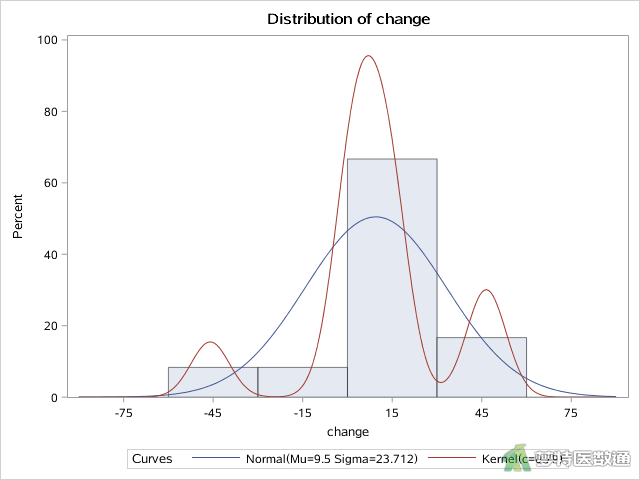
HISTOGRAM语句可以针对指定变量“Change”绘制直方图,该语句中NORMAL选项可作正态分布的密度曲线,而KERNERL选项可作基于数据的核分布密度曲线,可直观展示变量的分布情况,如图5所示。
2. 结果解读
图4显示了多种正态性检验的结果,Kolmogorov-Smirnov (柯尔莫哥洛夫-斯米诺夫,K-S)检验和Shapiro-Wilk (夏皮罗-威尔克正态性,S-W)检验为常用的正态性检验。K-S检验适用于大样本资料,本案查看S-W检验结果,结果显示P=0.0629<0.1,提示数据不服从正态分布;图2 的Q-Q图上散点与对角线的分布重合度较低,且图4 中数据的核分布密度曲线与正态分布密度曲线重合度较低,也可以认为数据不服从正态分布。所以本案例宜选用配对样本Wilcoxon符号秩检验。关于正态性检验的注意事项详见推文医学统计学核心概念及重要假设检验的软件实现(2/4)——正态性假设检验的SPSS实现。
(二) 统计描述及推断
1. 统计描述
(1) 软件操作
通过UNIVARIATE过程可分别得出原方法和新方法测得的血清谷草转氨酶含量的统计学描述信息:
proc univariate data= mydata.example plot normal; var old new; Histogram /normal kernel; run;
另外,两组差值“change”的统计学描述信息可由上一步骤中的UNIVARIATE过程给出:
proc univariate data= mydata.example plot normal; var change; Histogram /normal kernel; run;
(2) 结果解读
图6和图7分别为旧方法和新方法“Basic Statistical Measures (基本统计描述)”结果,可见使用原方法和新方法的两组“N (样本量)”均为12,原方法测得的血清谷草转氨酶含量“Mean(均值)”为143.42 nmol/SL,“Std. Deviation(标准差)”为80.02 nmol/SL,“Median(中位数)”为166.00 nmol/SL,“Interquartile Range (四分位间距)”为139.0 nmol/SL;新方法测得的血清谷草转氨酶含量“Mean(均值)”为152.92 nmol/SL,“Std. Deviation(标准差)”为80.62 nmol/SL,“Median(中位数)”为171.00 nmol/SL,“Interquartile Range (四分位间距)”为152.0 nmol/SL。从均值和中位数看两组治疗前后可能存在差异,但还需要依据统计学检验的结果进行判断。
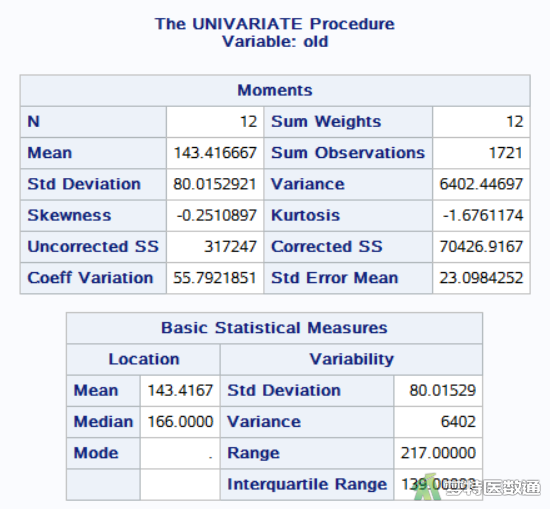

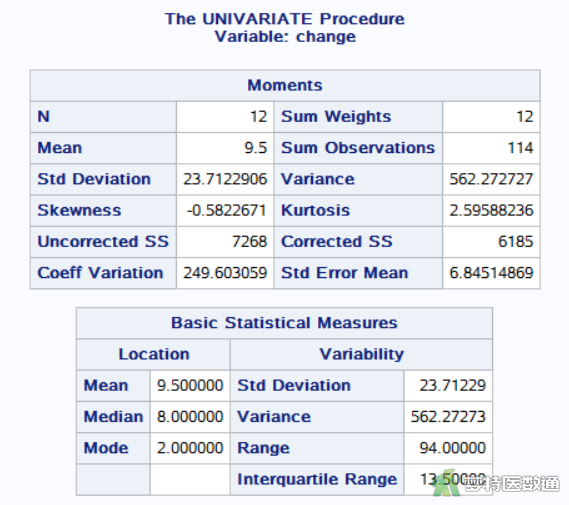
图8为新变量“change”的“Basic Statistical Measures (基本统计描述)”结果。可见新方法与原方法的差值的“Mean(均值)”为9.50 nmol/SL,“Std. Deviation(标准差)”为23.71 nmol/SL,“Median(中位数)”为8.00 nmol/SL,“Interquartile Range (四分位间距)”为13.5 nmol/SL。;由正态性检验结果可知,该数据不服从正态分布,所以采用中位数(四分位间距)描述数据,可将该差值描述为8.00 (13.50) nmol/SL。
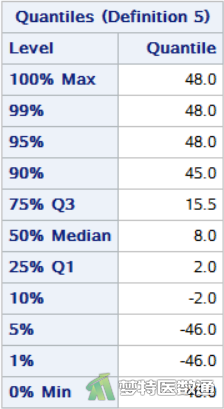
另外,该步骤还给出该差值数据的“Min(最小值)”、“Max(最大值)”、“25% Q1(第1四分位数)”、50% Median(中位数)和“75% Q3(第3四分位数)”等结果,如图9所示,故而该差值也可表示为8.00 (Q1~Q3:2.0~15.5) nmol/SL。
2. 统计学推断
(1) 软件操作
UNIVARIATE过程即可得到检验结果:
proc univariate data= mydata.example plot normal; var change; Histogram /normal kernel; run;
该过程默认指定了位置检验中原假设的总体位置的参数值为0, 即比较两组差值是否有统计学意义。如想设定另外的参数值,即可添加选项“Mu0=”。
注意:此处Mu0后为数字“0”,而不是英文字母“O”。
(2) 结果解读
由统计学描述信息可知,本案例中新方法测得的血清谷草转氨酶含量与原方法相比,其差值中位数为8.00 (13.50) nmol/SL,能够粗略看出两组之间存在差异,但究竟有无统计学意义还要看统计检验结果。
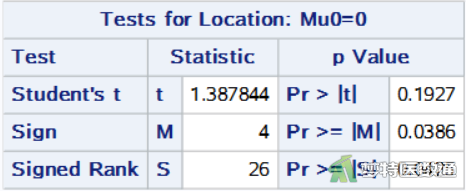
图10给出了“Tests for Location (位置检验)”的三种不同方法和结果。由于两组差值为非正态分布,参考Wilcoxon符号秩检验,即“Signed Rank (符号秩检验)”结果,检验统计量S =26, P= 0.0425 < 0.05,差异有统计学意义,故可以认为新方法测得的血清谷草转氨酶含量与原方法有差异。
注意:SAS中“Signed Rank (符号秩检验)”所使用的公式为秩次的绝对值之和减去n(n+1)/4,其中n为差值不等于0的样本量,故而此处减去12 (12+1)/4,所得统计量为S =26。而SPSS中给出的是负秩次的绝对值,同时给出标准化统计量Z,所用公式为Z =(秩和-n (n+1)/4) /标准误,具体数值可参考前期推文。
四、结论
本研究欲比较新法与原法检测血清谷草转氨酶含量是否存在差异,对新法与原法检测数值的差值进行正态性检验发现差值不服从正态分布,故本案例宜选用配对样本Wilcoxon符号秩检验。
结果显示,原法检测血清谷草转氨酶含量的中位数为166.00 (IQR:139.0) nmol/SL,新法检测血清谷草转氨酶含量的中位数为171.00 (IQR:152.0) nmol/SL,两种方法检测结果差值的中位数为8.00 (13.75) nmol/SL,差异有统计学意义(S =26, P= 0.0425 < 0.05),可以认为两种方法检测的结果不同。