局部加权线性回归(locally weighted linear regression),也称局部加权回归(lowess),为非参数回归方法,对数据分布、残差齐性没有严格要求,适合对线性和非线性关系的数据进行回归拟合。本文实例介绍简单局部加权回归在R软件中的实现步骤。
关键词:R软件;局部加权线性回归; 局部加权回归; loess; lowess
普通线性回归通常是基于最小二乘法(OLS)进行计算,需要数据满足残差正态、齐性等条件。但实际应用中,数据有时并不满足普通线性回归的前提条件,或者会出现严重欠拟合现象,此时可使用局部加权回归(lowess)进行数据分析。
一、案例介绍
为探究某中药经典名方在降血压方面的治疗效果,临床入组100例患者,采集在服用不同剂量下患者的收缩压数据,因变量为收缩压(SBP),自变量为每日服用剂量(dosage)。案例可从“附件下载”处下载。
二、问题分析
本案例研究某中药经典名方在降血压方面的治疗效果,因变量为连续型数据,考虑采用回归分析探究因变量与自变量之间的关系。经线性回归分析后发现,无论是在模型拟合程度、残差标准误等方面均不是很理想,考虑使用局部加权线性回归进行分析(见下文分析)。局部加权线性回归需满足以下3个条件:
条件1:因变量为连续型变量。本案例中因变量SBP为连续型变量,符合要求。
条件2:各观测值之间相互独立。该条件可通过软件分析后辅助判断。
条件3:自变量不存在显著异常值。该条件通过软件分析后判断。
三、软件操作及结果解读
(一) 导入数据
data <- read.csv('局部加权线性回归.csv') #导入数据
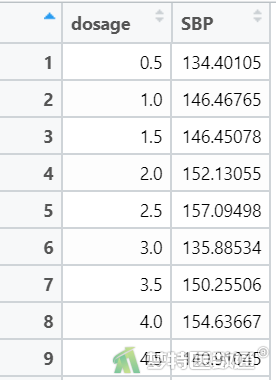
View(data) #查看数据(图1)
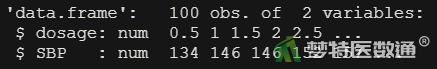
str(data) # 查看数据框结构(图2)
(二) 绘制散点图
绘制因变量与自变量的散点图。
library(ggplot2) ggplot(data,aes(x=dosage,y=SBP))+geom_point(size=3)+theme(axis.title.x =element_text(size=20), axis.title.y=element_text(size=20))+theme(axis.text.x =element_text(size=20), axis.text.y=element_text(size=20))
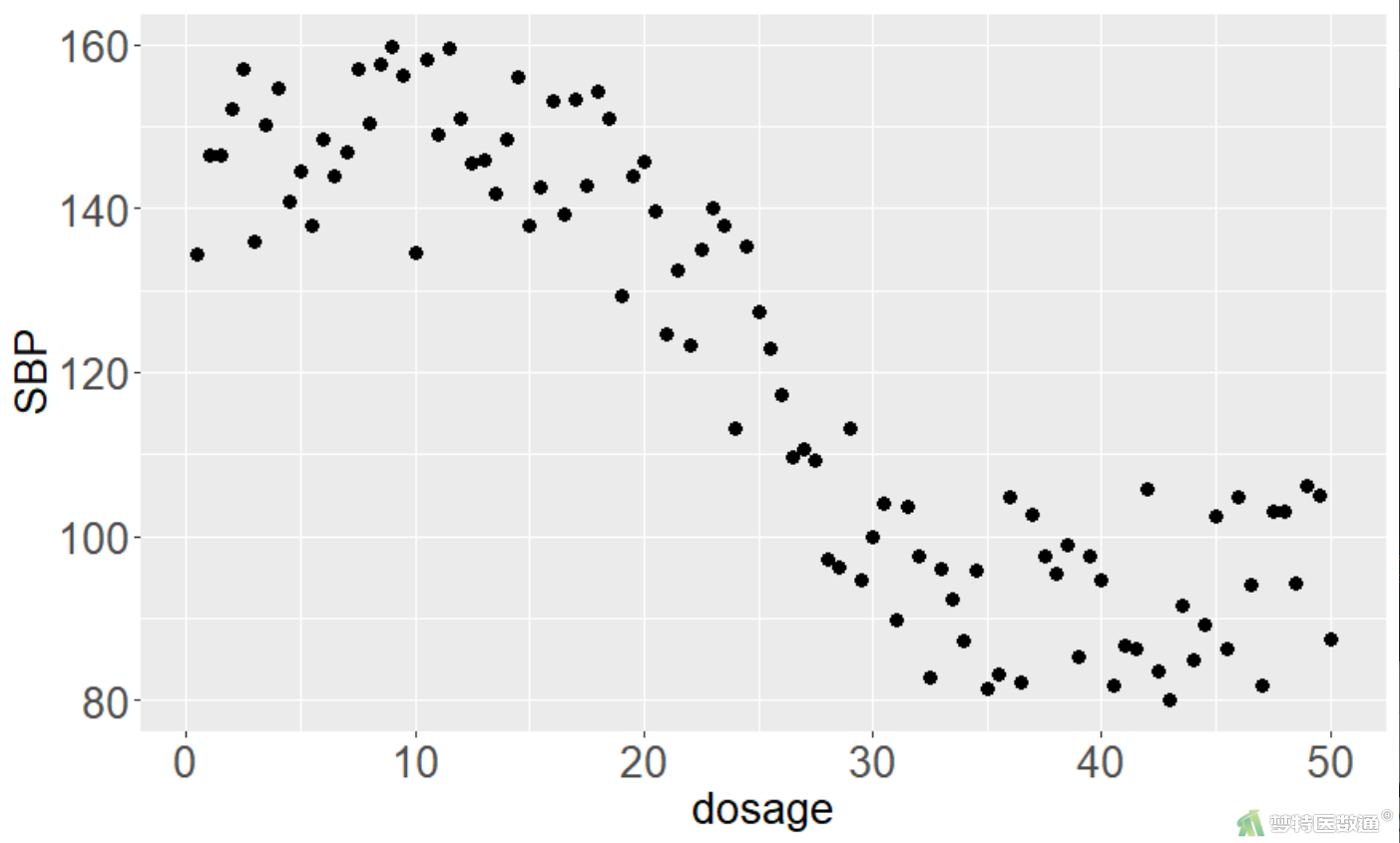
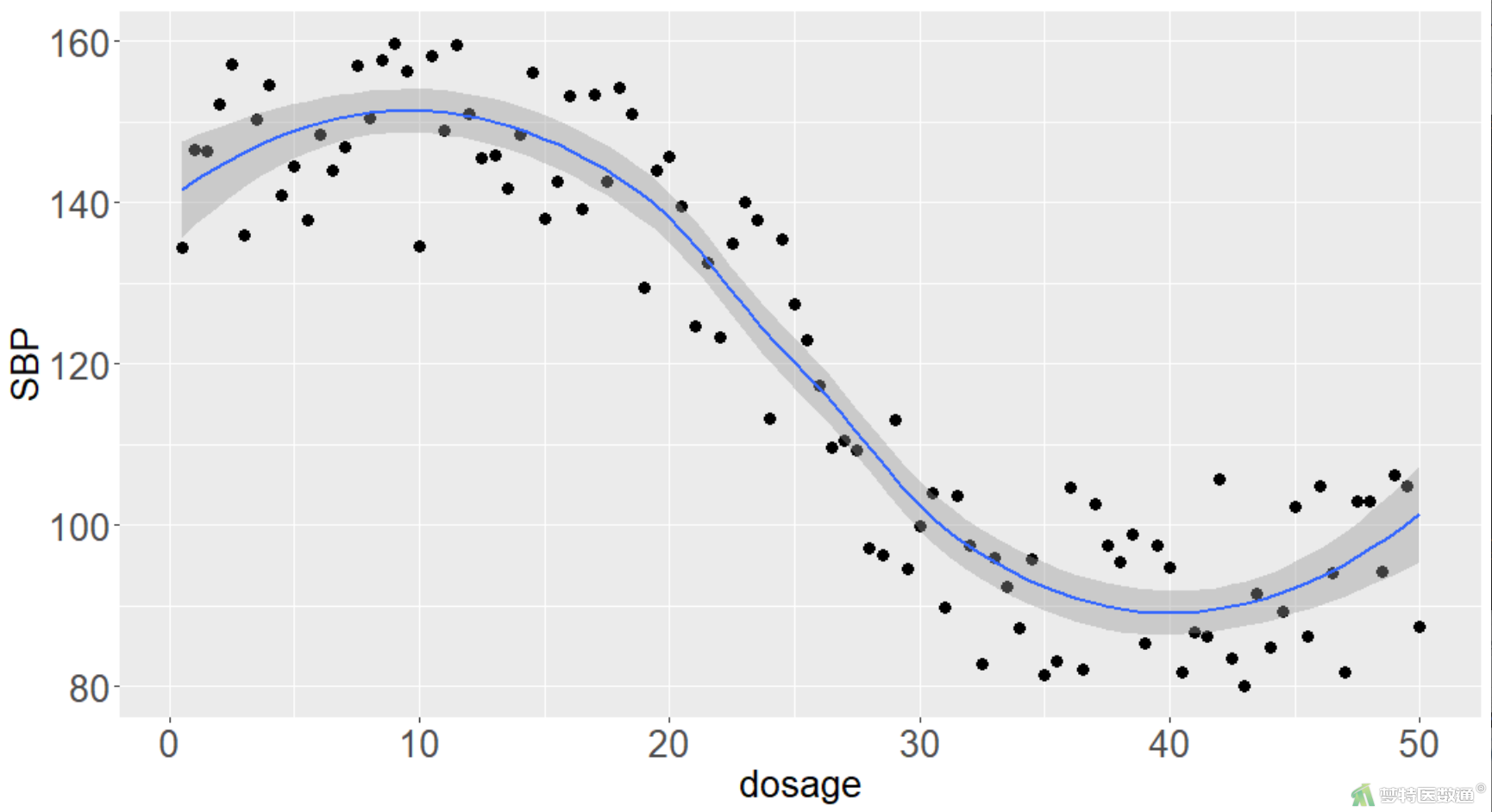
由图3可知自变量与因变量之间呈现倒S形的曲线回归。
(三) 线性回归
1. 软件操作
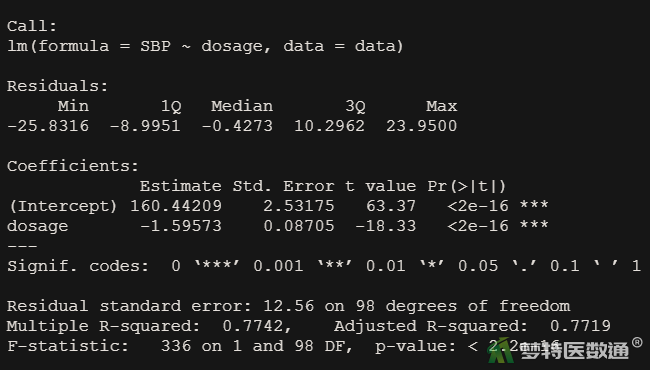
若使用lm函数进行线性回归拟合(图4),并绘制回归曲线(图5),结果见下文。
##线性回归拟合## lmfit <- lm(SBP~dosage,data = data) summary(lmfit)
##查看回归拟合曲线##
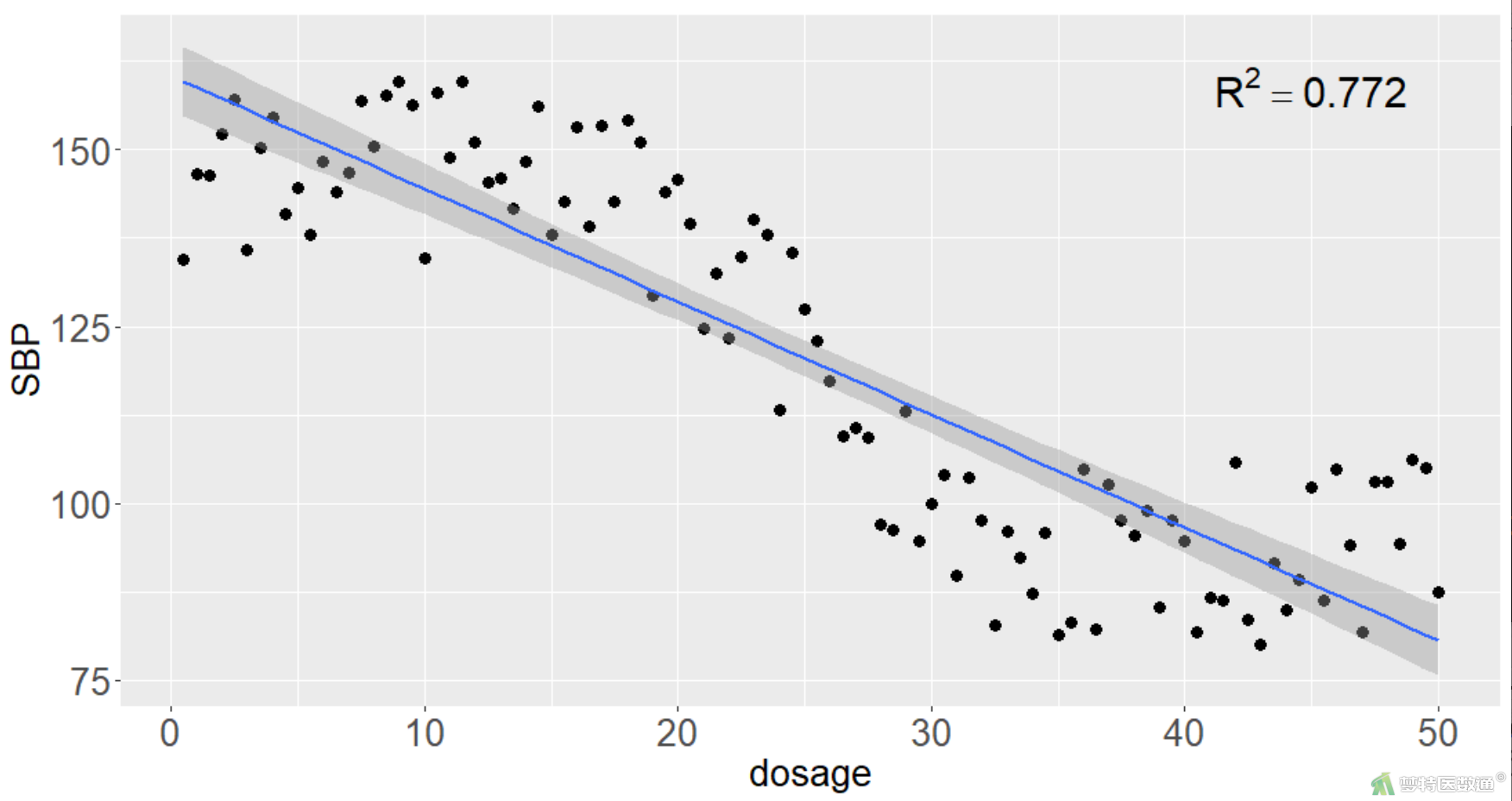
ggplot(data,aes(x=dosage,y=SBP))+geom_point(size=3)+stat_smooth(method = 'lm',se = T)+annotate("text",x=45,y=155,label="atop(R^2==0.772)",size = 8,parse=T)+theme(axis.title.x =element_text(size=20), axis.title.y=element_text(size=20))+theme(axis.text.x =element_text(size=20), axis.text.y=element_text(size=20))
2. 结果解读
从图4线性回归拟合结果可以看出,模型残差标准误(residual standard error)为12.56,调整后R2为0.772;从图5拟合曲线可以看出,不少数据存在偏差,模型整体拟合情况一般。可尝试使用局部加权线性回归进行回归模型拟合。
(三) 局部加权回归
1. 软件操作
(1) 初始模型拟合
使用loess函数实现局部加权回归,在局部加权回归中,比较重要的参数为数据子集的获取范围(即span,数值越大表明选择的数据子集越多,默认值为0.75)。
##初始模型拟合## loessfit <- loess(SBP ~ dosage,data = data,span=0.75) summary(loessfit)
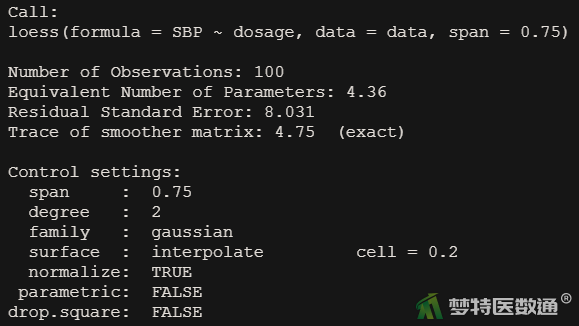
从图6可知,使用局部加权线性回归分析,模型残差标准误为8.031,比线性回归明显降低。
##绘制模型拟合曲线##(结果见图7) ggplot(data,aes(x=dosage,y=SBP))+geom_point(size=3)+stat_smooth(method = 'loess',formula=y~x,span=0.75,se = T)+theme(axis.title.x =element_text(size=20), axis.title.y=element_text(size=20))+theme(axis.text.x =element_text(size=20), axis.text.y=element_text(size=20))
(2) span值的选择
span表示数据子集的获取范围,数值越大表明选择的数据子集越多,默认为0.75,但默认值并不一定是模型最优取值。通常情况下采用交叉验证估计获取模型最优span取值。判断标准采用GCV准则,计算公式如下:
\(G C V=\frac{1}{n} \frac{\sum_{i=1}^{n}\left(y_{i}-\widehat{y}_{l}\right)^{2}}{\left(1-\frac{v_{i}}{n}\right)^{2}}\)
其中n为样本量,\(y_{i}\)为第i个真实值,\(\widehat{y}_{i}\)为第i个预测值,\(v_{i}\)为帽子矩阵的迹。
loess_loss <- function(s){
model_fit <- loess(formula = SBP~dosage,data = data,span=s)
gcv <- mean((model_fit$fitted-data$SBP)^2)/((1-model_fit$trace.hat/(length(data$SBP)))^2)
return(gcv)
} #定义-gcv准则计算函数
optim <- optimize(loess_loss, interval = c(0.01, 99)) #定义优化器
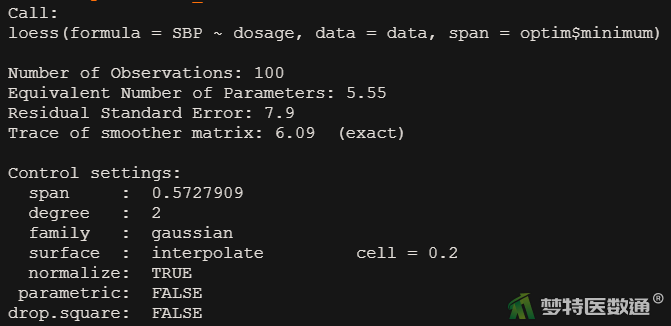
print(optim) #查看结果(图8)
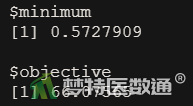
其中optim$minimum为span最佳取值,这里为0.5727909。
(3) 最佳模型
根据最佳span值,重新拟合模型(图9),结果见图10。
loessfit_best <- loess(SBP ~ dosage,data = data,span=optim$minimum) summary(loessfit_best)
##绘制拟合曲线## ggplot(data,aes(x=dosage,y=SBP))+geom_point(size=3)+stat_smooth(method = 'loess',formula=y~x,span=optim$minimum,se = T)+theme(axis.title.x =element_text(size=20), axis.title.y=element_text(size=20))+theme(axis.text.x =element_text(size=20), axis.text.y=element_text(size=20))
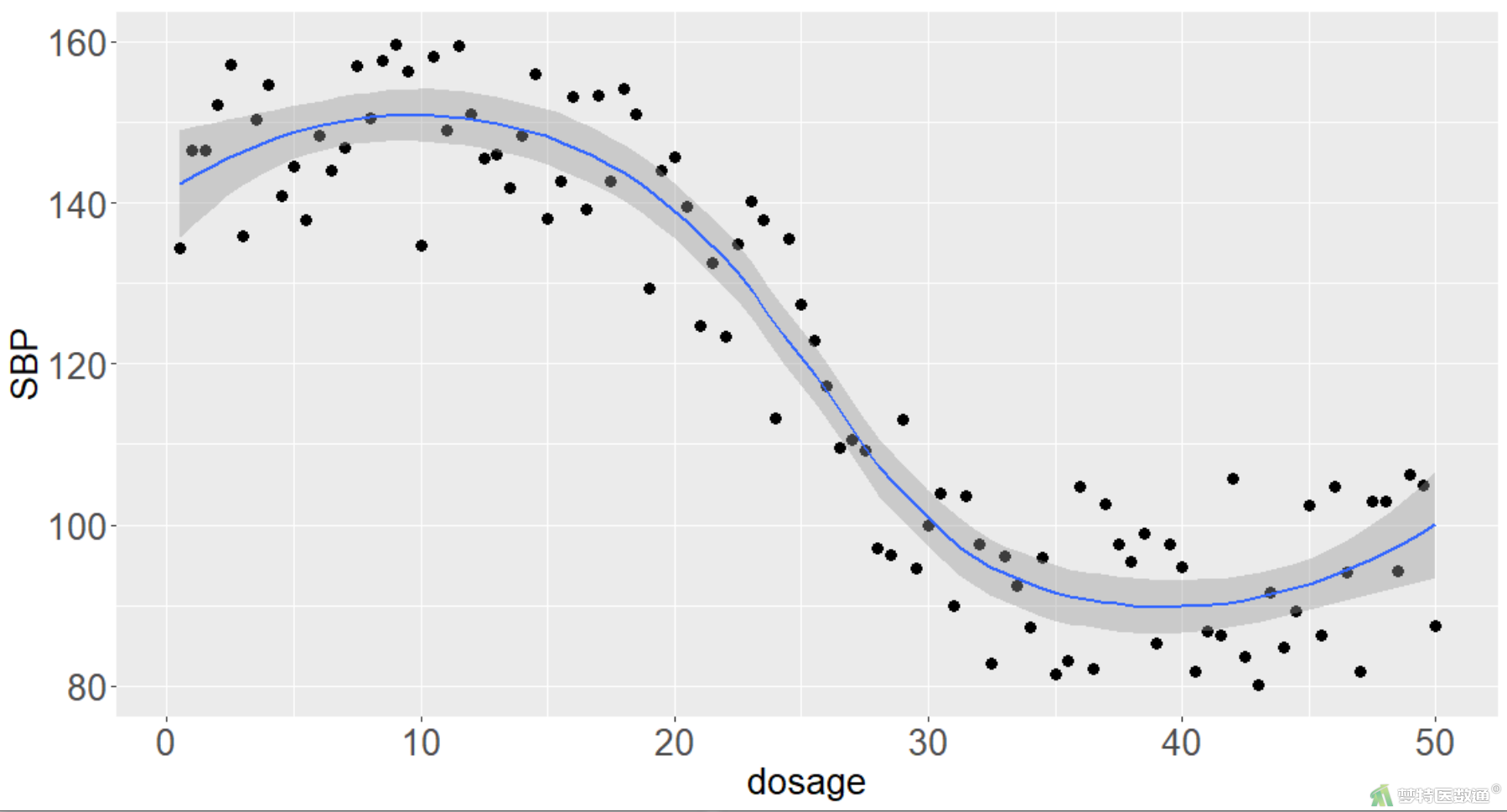
从图9可知,与最佳模型和初始模型比较,模型残差标准误从8.031进一步降低至7.9。
2. 适用条件判断
(1) 独立性考察
Durbin-Watson检验通常用来检测残差是否存在自相关,Durbin-Watson检验值分布在0~4之间,越接近2,观测值相互独立的可能性越大。需要注意的是,判断观测值是否独立,主要取决于研究设计和数据收集阶段的质量控制,Durbin-Watson检验一般用于辅助判断。
① 软件操作
library(car)
model_best = loess(SBP ~ dosage,data = data,span=0.57279087929112)
print(sprintf('Durbin-Watson检验值:%s',durbinWatsonTest(model = model_best$residuals)))
② 结果解读

从图 11可知,DW Statistic为2.312,说明观测值相互独立,满足条件2。
(2) 异常值考察
① 软件操作
##绘制两组变量箱型图##
data_tab = data.frame(group = c(rep('SBP',each=nrow(data)),rep('dosage',each=nrow(data))),values = c(data$SBP,data$dosage))
labels <- c("SBP","dosage") #设置分组标签
boxplot(data_tab$value ~ data_tab$group, names = labels,
xlab = "group", ylab = 'values') #分组绘制箱线图
② 结果解读
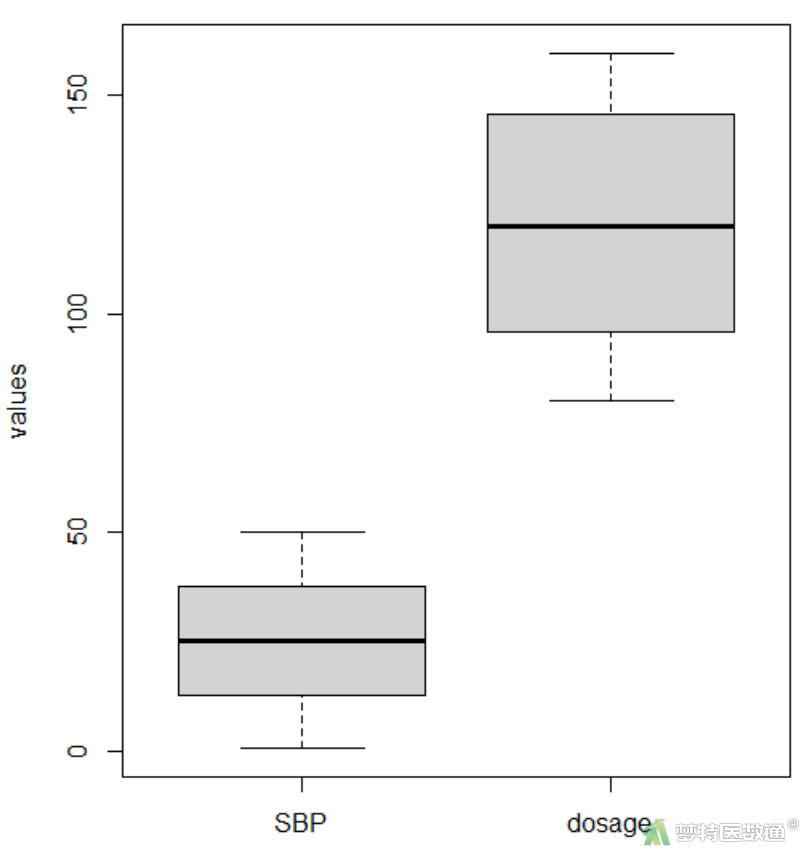
从图 12可知,本案例数据未发现明显异常值,满足条件3。
3. 结果解读
从简单局部加权线性回归拟合结果可以看出,模型残差标准误(residual standard error)为7.9,相比线性回归模型残差标准误12.56明显下降;从拟合曲线可以看出,模型整体拟合效果良好,且表明某中药经典名方的服用剂量与降血压效果呈现S型关系。