在前面文章中我们介绍了两因素重复测量方差分析(Two-way Repeated-Measures ANOVA)的假设检验理论,本篇文章将实例演示在SAS软件中实现两因素重复测量方差分析——不存在交互作用时的操作步骤。
关键词:SAS; 重复测量; 重复测量资料; 重复测量方差分析; 两因素重复测量方差分析; 球形检验; 交互作用; 主效应; 单独效应
一、案例介绍
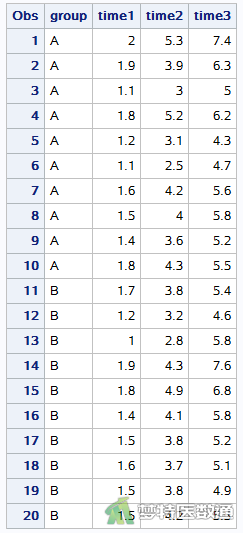
研究A、B两种饲料对家兔的增重效果,选择20只家兔,随机分成两组,第一组用饲料A饲养,第二组用饲料B饲养,并于试验开始后第一个月(time1)、第二个月(time2)、第三个月(time3)分别测量2组家兔体重,试比较A、B两种饲料对家兔的增重效果有无差别?数据见图1。本文案例可从“附件下载”处下载。
二、问题分析
本案例的分析目的是比较A、B两种饲料对家兔的增重效果有无差别。由于3个时间点的数据属于重复测量数据,且有两个组别,可以使用两因素(时间因素time和分组因素group)重复测量方差分析。但需要满足以下6个条件:
条件1:观察变量唯一,且为连续变量。本研究中观察变量只有体重,且为连续变量,该条件满足。
条件2:有两个分析因素。本研究有时间因素time和分组因素group两个因素,该条件满足。
条件3:观察变量为重复测量数据,即不满足独立性。本研究中两个组别在3个时间点时测量的体重均是针对同一批样本,因此不满足独立性,该条件满足。
条件4:观察变量不存在显著的异常值,该条件需要通过软件分析后判断。
条件5:各组、各水平(时间点)观察变量为正态(或近似正态)分布,该条件需要通过软件分析后判断。
条件6:相互比较的各处理水平(组别)的总体方差齐,该条件需要通过软件分析后判断。
三、软件操作及结果解读
(一) 导入数据
①利用LIBNAME语句建立SAS逻辑库关联,注意逻辑库名称要求,即最大长度8字符,必须以字母或下划线“_”开始,可以是字母、数字和下划线的任意组合。具体代码如下:
libname mydata ‘D:\mydata’;
通过这一步骤,SAS能够识别引号中的物理位置,将逻辑库建立在该目录下,同时在以下过程中新建的SAS表格便可以永久储存在该位置,便于反复读取和使用。先运行该代码使其生效。
②利用PROC IMPORT语句导入文件,代码如下:
proc import out= mydata.example
datafile=" D:\mydata\两因素重复测量方差分析一.csv"
dbms=csv replace;
getnames=yes;
run;
该过程在mydata逻辑库中生成example数据集,数据文件由DATAFILE=选项指定,DBMS=选项指定其数据库类型。该案例中初始数据集为csv文件,故而使用“dbms=csv”指定。如果已经存在相同名称的SAS数据集,即可使用REPLACE选项进行覆盖。GERNAMES=YES选项指定从第2行开始读取数据,将数据集的首行变量名作为SAS数据集的变量名。
(二) 适用条件判断
1. 条件4判断(异常值判断)
(1) 软件操作
运用UNIVARIATE过程进行检验:
proc univariate data= mydata.example plot; var time1 time2 time3; by group; run;
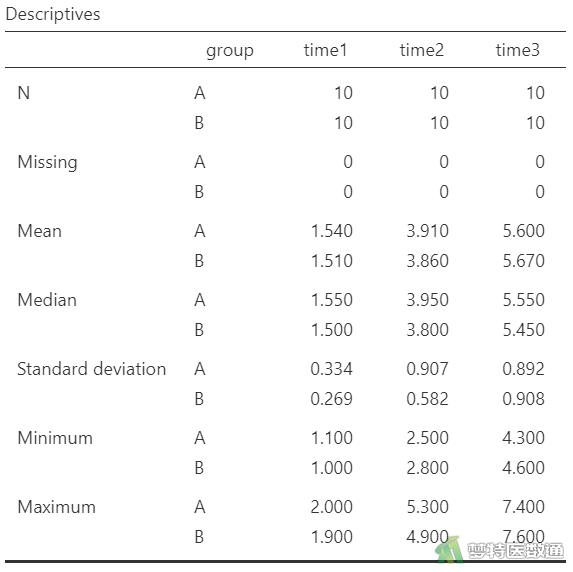
其中“mydata.example”为上一步中导入的数据集名称,“time1”、“time2”、“time3”为分析变量名,即分别为3个时间点检测的家兔体重。“by group”表示分A、B两组分别对三个时间点的家兔体重进行异常值判断。因此将会出现6个检验结果,总结如图2:
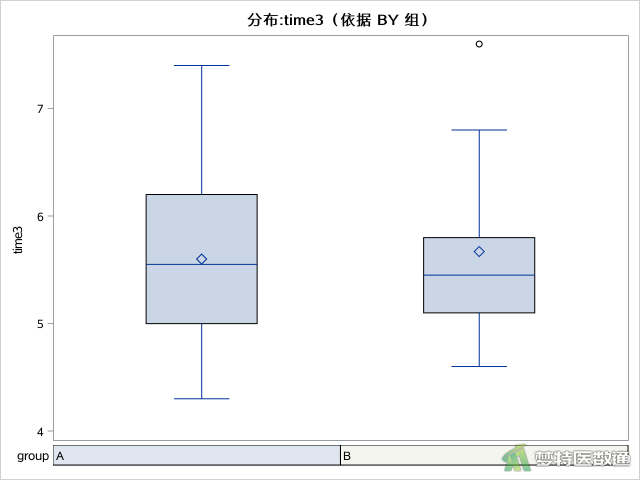
描述数据分布情况的箱线图,见图3-1—图3-3所示。
(2) 结果解读
图2“Descriptives (描述性分析)”表格中,总结出了各组观察变量的最小值和最大值,依据专业尚不能认为存在异常值;此外,图3-1—图3-3中的箱线图也未提示任何异常值。综上,本案例未发现需要删除的异常值,满足条件4。
2. 条件5判断(正态性检验)
(1) 软件操作
运用UNIVARIATE 过程对两组各时间点的家兔体重进行正态性检验,示例代码如下:
proc univariate data= mydata.example normal; var time1 time2 time3; by group; run;
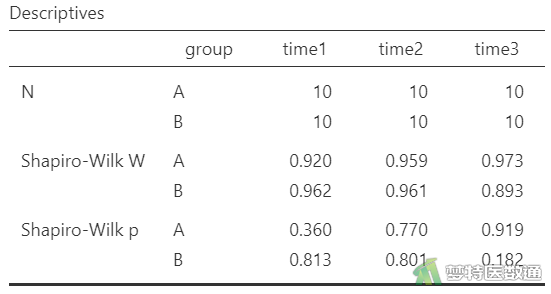
两因素重复测量方差分析时,需要分别考察每一组的正态性情况,因此将会出现6种检验结果,总结如图4:
(2) 结果解读
图4的正态性检验结果显示A组3个时间点P=0.360、0.770、0.919,B组三个时间点P=0.813、0.801、0.182,均>0.1,提示各组数据均服从正态分布。此外,本案例也可以绘制Q-Q图,结果也提示各组数据均服从正态分布(请读者自行操作)。综上,本案例满足条件5。关于正态性检验的注意事项详见文章(医学统计学核心概念及重要假设检验的软件实现(2/4)——正态性假设检验的SPSS实现)。
3. 条件6判断(方差齐性检验)
(1) 软件操作
proc anova data=mydata.example; class group; model time1 time2 time3=group; means group / hovtest=levene(type=abs); run;
(2) 结果解读
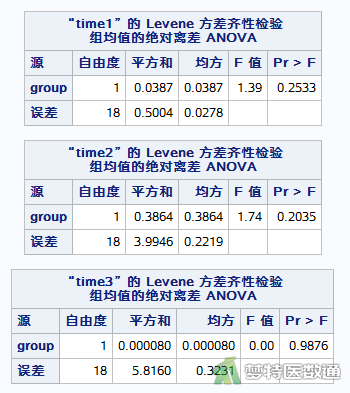
图5 “Homogeneity of Variances Test (Levene’s) (Levene’s方差齐性检验)”表格显示,time1、time2、time3时,两组之间的方差齐性检验结果分别为F=1.39、P=0.2533,F=1.74、P=0.2035,F=0.00、P=0.9876;提示每个时间点的两组之间都满足方差齐性。本案例满足条件6。关于方差齐性检验的更多内容请阅读(医学统计学核心概念及重要假设检验的软件实现(4/4)——方差齐性检验及SPSS实现)。
(三) 球形假设检验
1. 软件操作
proc glm data=mydata.example;
class group;
model time1 time2 time3=group;
repeated time 3/printe;
run;
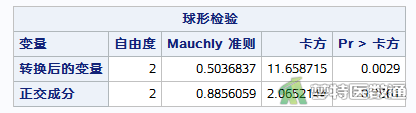
通过printe进行球形检验,结果如图6中“正交成分”所示。
2. 结果解读
由图6的球形检验结果可知,W=0.886,P=0.3561,表示满足球形假设。因此本案例可以直接采用非校正方法分析的结果。
(四) 统计描述
1. 软件操作
proc glm data=mydata.example;
class group;
model time1 time2 time3 = group;
repeated time 3 ;
lsmeans group/out=means;
run;
proc sort data=means;by group _name_;
proc print;run;
proc glm data=mydata.example;
class group;
model time1-time3 = group;
output out=out lclm=time1-time3 uclm=time1-time3;
run;
proc print data=out;run;
data out;
set out;
length time_1 $25 time_2 $25 time_3 $25;
time_1=compress(put(time12,6.3)) || '-' || compress(put(time13,6.3));
time_2=compress(put(time22,6.3)) || '-' || compress(put(time23,6.3));
time_3=compress(put(time32,6.3)) || '-' || compress(put(time33,6.3));
proc print;run;
proc transpose data=out out=out1(drop=ci2-ci10) prefix=CI;
var time_1-time_3;
by group;
proc print;run;
proc sort data=out1;
by group _name_;
proc print;run;
data a;
merge out1 means;
CI=CI1;
label group='分组' _NAME_='时间点' CI='置信区间' LSMEAN='均值' STDERR='标准误';
drop CI1;
proc print label;run;
proc transpose data=out out=out2(keep = group _NAME_ _LABEL_ CI1) prefix=CI;
var time12 time22 time32;
by group;
proc print;run;
proc transpose data=out out=out3(keep = group _NAME_ _LABEL_ CI2) prefix=CI;
var time13 time23 time33 ;
by group;
proc print;run;
data combine(rename=(LSMEAN=MEAN));
merge out2 out3 means;
drop _NAME_;
proc print;run;
proc sgplot data=combine;
scatter x=_label_ y=mean/ yerrorupper=CI2 yerrorlower=CI1 group=group groupdisplay=cluster clusterwidth=0.2;
series x=_label_ y=mean/group=group groupdisplay=cluster clusterwidth=0.2;
run;
2. 结果解读
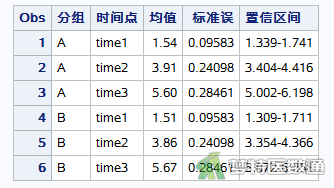
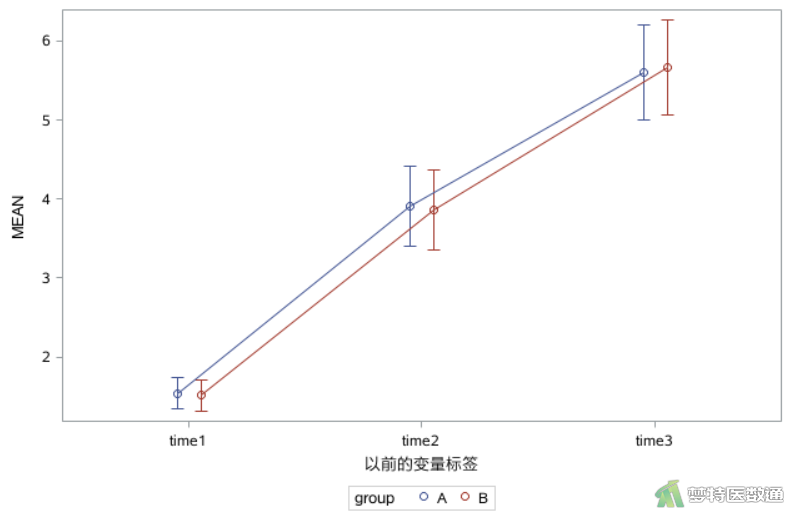
为了结果解释方便,首先在图7中总结了A、B两组在3个时间点的均值和标准差及其95%置信区间,图8则直观展示了两组的变化趋势。图7列出了A、B两组time1、time2、time3时间点的均值分别为1.54、3.91、5.60 kg和1.51、3.86、5.67 kg。图8绘制了两组三个时间点体重的变化情况,可见两组的体重均有增加,并且增加的幅度基本保持一致。
(五) 交互作用判断
由于本案例有两个因素(一个为时间因素time,另一个为分组因素group),因此需要首先判断两个因素之间是否存在交互作用。如果交互作用有统计学意义,则需要分析单独效应。
1. 软件操作
proc glm data=mydata.example;
class group;
model time1 time2 time3=group/nouni;
repeated time 3(0 1 2) contrast(1)/printe summary;
run;
2. 结果解读
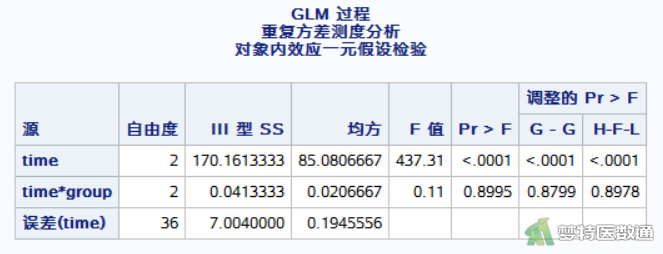
图9显示,time与group之间的交互作用结果为Ftime*group=0.11,P=0.8995,提示time与group之间的交互作用无统计学意义。因此,本案例可直接以主效应分析结果进行判断,如果交互作用有统计学意义,则需要分析单独效应。
(六) 时间效应分析
以“Within Subjects Effects (主体内对比效应)”中time的检验结果可知,Ftime=437.308,P<0.001(图9),认为体重变化具有时间变化趋势。
(七) 组间效应分析
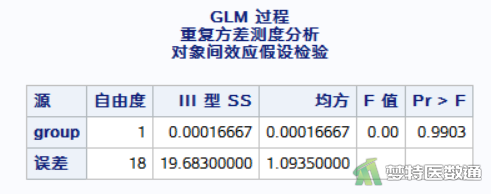
以图10中group的检验结果可知,Fgroup<0.001,P=0.9903,表示两种饲料对家兔的增重效果差异无统计学意义。
(八) 事后检验(两两比较)
上面分析得出了“体重变化具有时间趋势”的结论,即不同时间点体重差异有统计学意义,但具体是哪些时间点之间存在差异尚不清楚,因此需要进行事后检验,开展两两比较。
1. 软件操作
proc glm data=mydata.example;
class group;
model time1 time2 time3=group/nouni;
repeated time 3(0 1 2) contrast(1)/printe summary;
run;
程序中repeated time 3(0 1 2) contrast(1)/printe summary;可展示以time1为参照的两两比较结果,通过修改contrast(1)中的数字,可以改变参照组,进而得到不同时间点之间的两两比较结果,见图11-图12。
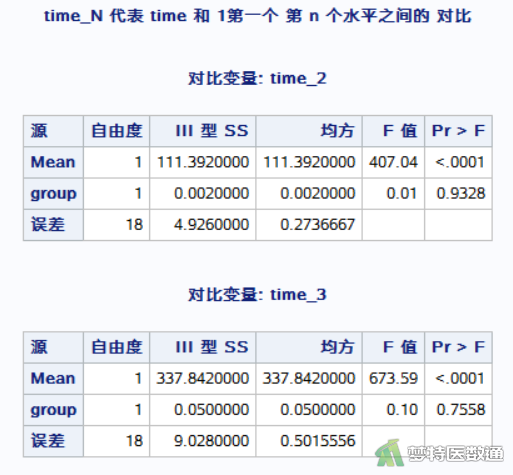
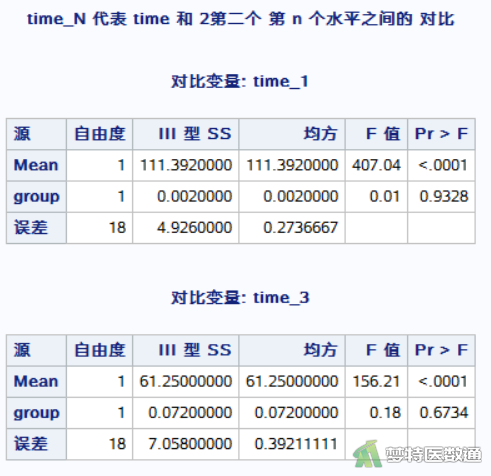
2. 结果解读
图11和图12提供了各时间点两两比较的 “df (自由度)”、“F (统计量F值)”、“P (未校正P值)”。可知,差异均有统计学意义(P<0.001)。
四、结论
本研究采用两因素重复测量方差分析比较A、B两种饲料对家兔的增重效果有无差别。通过专业知识判断,数据不存在异常值;通过Shapiro-Wilk检验,提示各组数据服从正态分布;通过Levene’s检验,提示每个时间点的两组之间都满足方差齐性;球形度检验提示,满足球形假设(W=0.886,P=0.3561);组别与time无交互作用(Ftime*group=0.11,P=0.8995),故进行主效应分析。
A、B两组在试验开始后第1个月、第2个月和第3个月时的体重分别为1.540±0.334、3.910±0.907、5.600±0.892 kg和1.510±0.269、3.860±0.582、5.670±0.908 kg。两因素重复测量方差分析结果显示,在试验开始后第一个月、第二个月和第三个月时2组家兔的体重均呈上升趋势(Ftime=437.31,P<0.001),但两种饲料对家兔的增重效果差异无统计学意义(Fgroup=0.00,P=0.9903)。进一步采用“Bonferroni”校正法进行两两比较可知,随着时间的延长,试验开始后第第2个月和第3个月与第1个月试验开始时相比,体重增加均有统计学意义(P<0.001)。综上,两种饲料对家兔的增重效果无差异。