在前面文章中我们介绍了单因素重复测量方差分析(One-Way Repeated Measures ANOVA)的假设检验理论,本篇文章将使用实例演示在R软件中实现单因素重复测量方差分析的操作步骤。
关键词:R语言; R软件; 重复测量; 重复测量资料; 重复测量方差分析; 单因素重复测量方差分析; 球形检验; 交互作用; 主效应; 单独效应
一、案例介绍
检验科研究血样放置时间对某生化指标浓度检测的影响,采集了10份人血标本,分别在放置0分钟(T0)、30分钟(T30)、60分钟(T60)和90分钟(T90)时对该指标的浓度(mmol/L)进行检测,分析放置时间是否对该生化指标检测结果有影响?部分数据见图1。本文案例可从“附件下载”处下载。
二、问题分析
本案例的分析目的是比较4个时间点的生化指标浓度是否有差异。由于4个时间点的数据属于重复测量数据,可以使用单因素重复测量方差分析(One-Way Repeated Measures ANOVA)。但需要满足4个条件:
条件1:观察变量唯一,且为连续变量。本研究中观察变量为生化指标浓度,且为连续变量,该条件满足。
条件2:观察变量为重复测量数据,即不满足独立性。本研究中4个时间点测量的生化指标浓度均是针对同一批样本,因此不满足独立性,该条件满足。
条件3:观察变量不存在显著的异常值,该条件需要通过软件分析后判断。
条件4:各水平(时间点)观察变量为正态(或近似正态)分布,该条件需要通过软件分析后判断。
三、软件操作及结果解读
(一) 导入数据
mydata <- read.csv("单因素重复测量方差分析.csv") #导入CSV数据
View(mydata) #查看数据
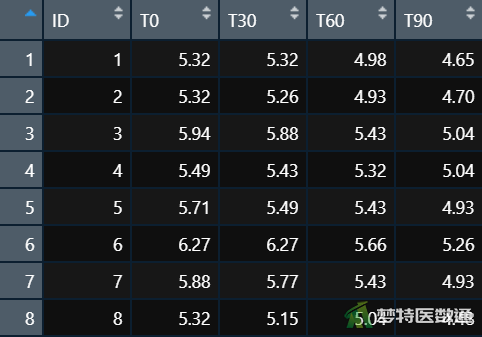
在数据栏目中可以查看全部数据情况,数据集中共有5个变量和10个观察数据,5个变量分别代表10个人的编号(ID)、放置0分钟(T0)、30分钟(T30)、60分钟(T60)和90分钟(T90)。
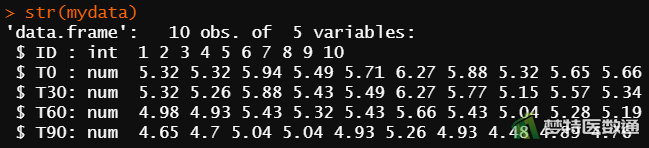
如果数据集较大也可使用如下命令查看数据框结构:
str(mydata) #查看数据框结构
(二) 适用条件判断
1. 条件3判断(异常值判断)
(1) 软件操作
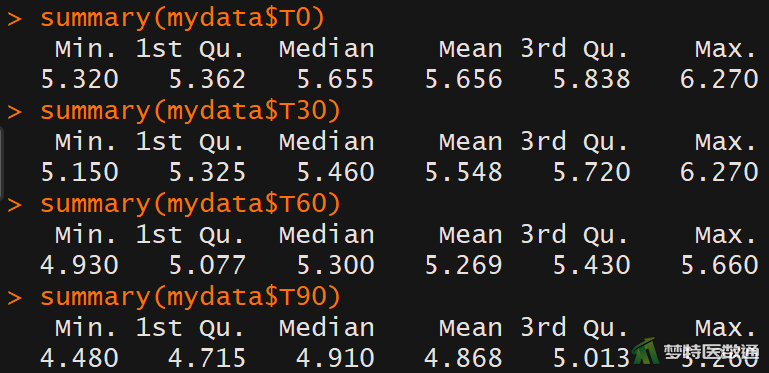
## 描述数据基本情况 ##
summary(mydata$T0) #描述T0的基本情况 summary(mydata$T30) #描述T30的基本情况 summary(mydata$T60) #描述T60的基本情况 summary(mydata$T90) #描述T90的基本情况
## 分组查看缺失值情况 ##
is.na(mydata$T0) #查看子集1小鼠体重有无缺失值 is.na(mydata$T30) #查看子集2小鼠体重有无缺失值 is.na(mydata$T60) #查看子集3小鼠体重有无缺失值 is.na(mydata$T90) #查看子集1小鼠进食量有无缺失值
## 分组绘制箱线图 ##
par(mfrow = c(1, 4)) #设置画1行4个图片
boxplot(mydata$T0, ylab = expression("T0")) #绘制T0箱线图
boxplot(mydata$T30, ylab = expression("T30")) #绘制T30箱线图
boxplot(mydata$T60, ylab = expression("T60")) #绘制T60箱线图
boxplot(mydata$T90, ylab = expression("T90")) #绘制T90箱线图
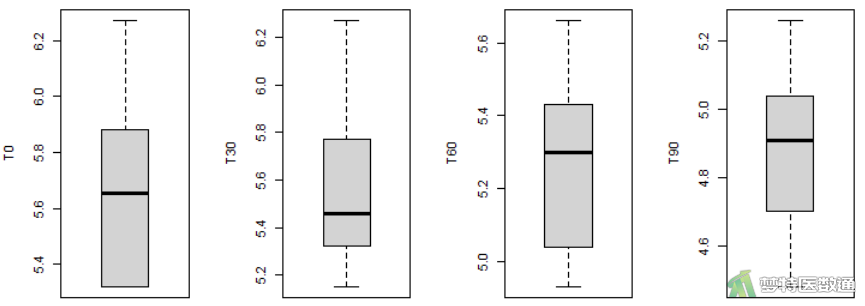
(2) 结果解读
图3“summary (描述性分析)”命令运行结果,列出了观察变量的“Min.(最小值)、“1st QU.(第一四分位数)”、“Median(中位数)”、“Mean(平均值)”、“3st QU.(第三四分位数)和“Max.(最大值)”,依据专业尚不能认为存在异常值的情况;此外,图5中的箱线图也未提示任何异常值。综上,本案例未发现需要处理的异常值,满足条件3。
2. 条件4判断(正态性检验)
(1) 软件操作
## 绘制Q-Q图 ##
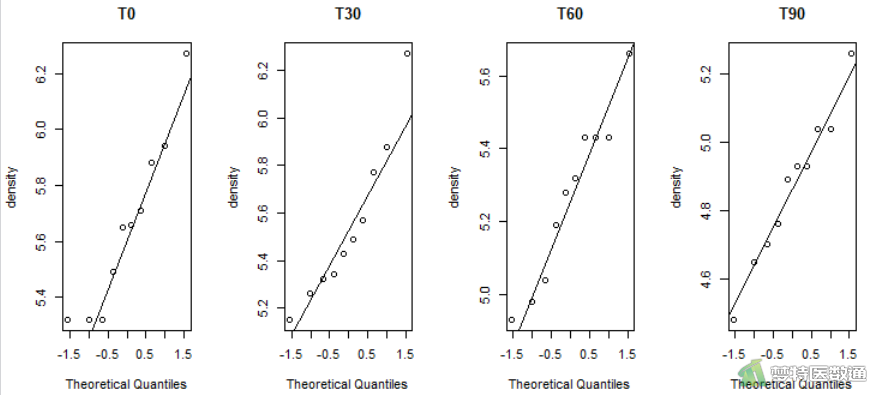
par(mfrow = c(1, 4)) #设置画1行4个图片 qqnorm(mydata$T0, ylab="density", main="T0") #绘制T0组的qq图 qqline(mydata$T0) #增加趋势线 qqnorm(mydata$T30, ylab="density", main="T30") #绘制T30的qq图 qqline(mydata$T30) #增加趋势线 qqnorm(mydata$T60, ylab="density", main="T60") #绘制T60的qq图 qqline(mydata$T60) #增加趋势线 qqnorm(mydata$T90, ylab="density", main="T90") #绘制T90的qq图 qqline(mydata$T90) #增加趋势线
## 正态性检验 ##
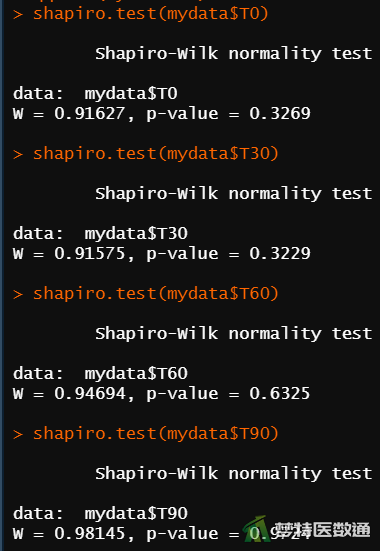
shapiro.test(mydata$T0) #检验T0的正态性 shapiro.test(mydata$T30) #检验T30的正态性 shapiro.test(mydata$T60) #检验T60的正态性 shapiro.test(mydata$T90) #检验T90的正态性
(2) 结果解读
图6的Q-Q图显示各组数据基本服从正态性,图7的正态性检验结果显示各时间点的P值分别为0.3269、0.3229、0.6325、0.9724,均>0.1,提示四组数据均服从正态分布。综上,本案例满足条件4。
(三) 球形假设检验
1. 软件操作
## 球形检验 ##
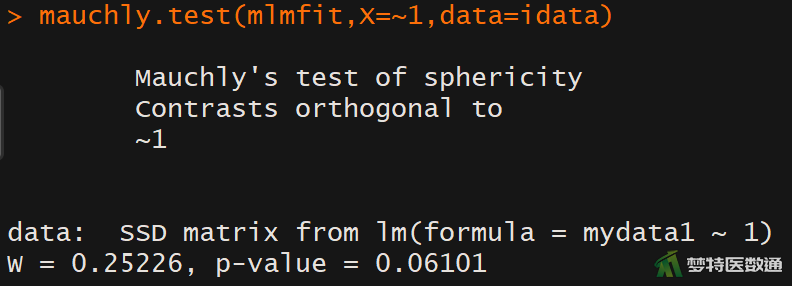
mydata1<-as.matrix(mydata[-1]) #数据整理为矩阵格式 mlmfit<-lm(mydata1~1) #计算回归系数等 idata <- data.frame(time = gl(4, 1, 4, labels = c(1,2,3,4))) #定义分组 mauchly.test(mlmfit,X=~1,data=idata) #球形检验
2. 结果解读
由图8的“Tests of Sphericity (球形度检验)”结果可知,W=0.252,P=0.061,表示满足球形假设。因此,本案例可以直接采用非校正方法分析的结果。
(四) 统计描述
1. 软件操作
## 计算标准差 ##
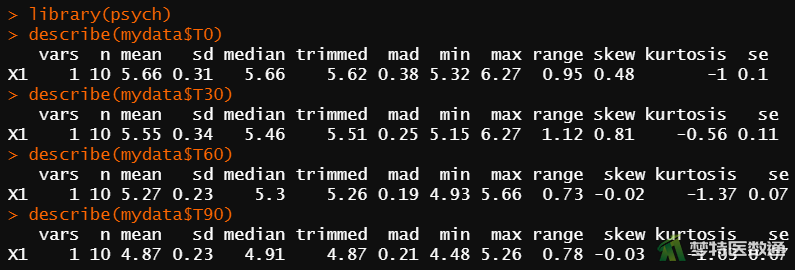
library(psych) #调用包“psych” describe(mydata$T0) #T0的描述统计 describe(mydata$T30) #T30的描述统计 describe(mydata$T60) #T60的描述统计 describe(mydata$T90) #T90的描述统计
## 宽数据转换为长数据 ##
library(reshape2) # 调用包“reshape2”
mydata_long<-melt(data=mydata,id.vars=c('ID'),#保留不变的变量
measure.vars=c('T0','T30','T60','T90'), #想要转换的变量
variable.name='time', #转换后的分类变量名
value.name='tvalue') #转换后的数值变量名
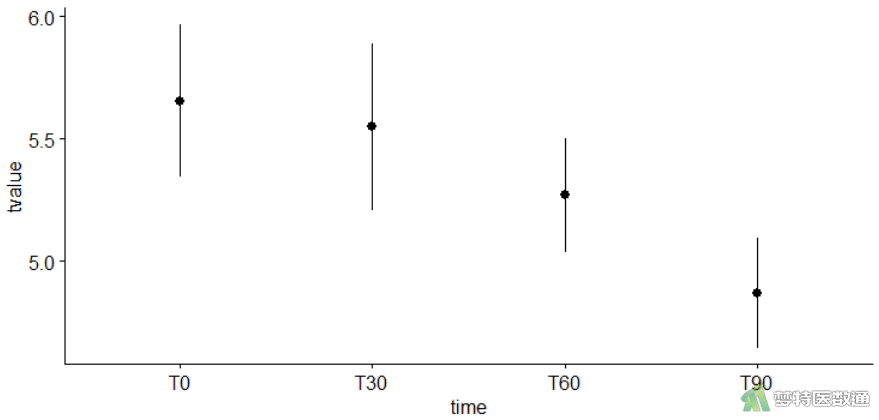
library(ggpubr) #调用包“ggpubr”
ggerrorplot(mydata_long, x = "time", y = "tvalue", #画平均值标准差图
desc_stat = "mean_sd")
2. 结果解读
图9列出了T0、T30、T60、T90时间点的均值分别为5.66±0.31、5.55±0.34、5.27±0.23、4.87±0.23 mmol/L。图10绘制了四个时间点该生化指标浓度的变化情况,可见随着放置时间的延长,浓度逐渐降低。
(五) 统计学推断
1. 软件操作
## 重复测量的方差分析 ##
install.packages("ez") #安装包“ez”
library(ez) #调用包“ez”
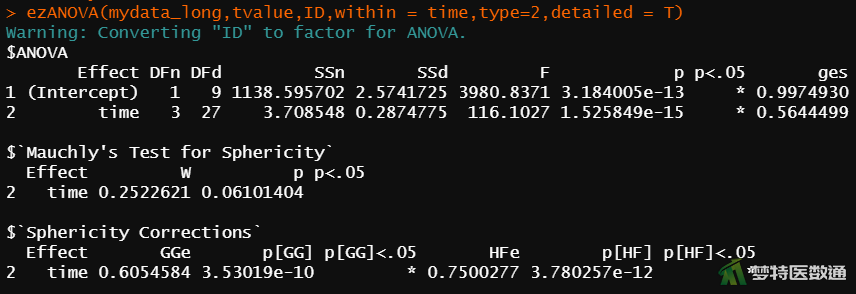
ezANOVA(mydata_long,tvalue,ID,within = time,type=2,detailed = T) #重复测量方差分析
2. 结果解读
图11重复测量方差分析结果显示,非校正法分析自由度为3,均方为3.709,F=116.1,P<0.001;提示不同时间点该生化指标浓度差异有统计学意义。
(六) 事后检验(两两比较)
上面分析得出了“不同时间点生化指标浓度差异有统计学意义”的结论,但是到底是哪些组别之间存在差异尚不清楚,因此需要进行事后检验,开展两两比较。
1. 软件操作
## 事后检验 ##
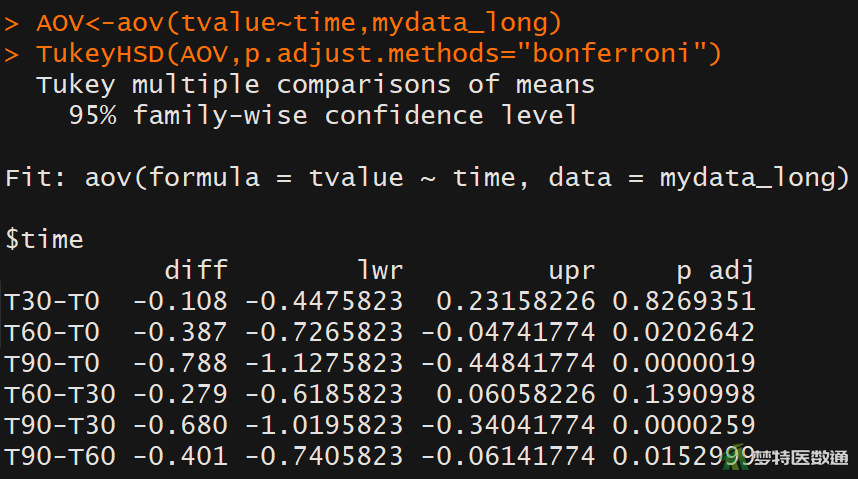
AOV<-aov(tvalue~time,mydata_long) #检验不同时间之间的差异 TukeyHSD(AOV,p.adjust.methods="bonferroni") #事后两两比较
2. 结果解读
“TukeyHSD (事后检验比较)”图12中提供了各组两两比较的“diff (均数差)”、 “lwr (95%置信区间下限)”、“upr (95%置信区间上限)”和“p.adj (调整后的P值)”。可知,随着时间的延长,T90及T60时刻与T0时刻相比,均值差逐渐增大,且均有统计学意义(P<0.05);T60及T30时刻与T90时刻相比,均值差增加,差异均有统计学意义(P<0.05)。
四、结论
本研究采用单因素重复测量方差分析判断四个时间点的生化指标浓度是否有差异。通过专业知识判断,数据不存在异常值;通过Shapiro-Wilk检验,提示各组数据服从正态分布;球形度检验提示满足球形假设(W=0.252,P=0.061),使用未校正法进行数据分析。
T0、T30、T60、T90时间点的生化指标浓度均值分别为5.66±0.31、5.55±0.34、5.27±0.23、4.87±0.23 mmol/L。分析提示,不同时间点生化指标浓度差异有统计学意义(F=116.1,P<0.001)。进一步采用“Bonferroni”校正法进行两两比较,可知随着时间的延长,T90及T60时刻与T0时刻相比,均值差逐渐增大,且均有统计学意义(P<0.05);T60及T30时刻与T90时刻相比,均值差增加,差异均有统计学意义(P<0.05)。综上可知,放置时间对该生化指标检测结果具有较大的影响。
五、知识小贴士
重复测量设计常见于同一组研究对象多次、多个部位或多个维度测量数据间的比较,包括时间重复测量、部位重复测量和维度重复测量。时间重复测量如,同一组患者用药后多个时间点某疗效指标的比较;部位重复测量如,相同研究对象,身体多个部位某指标(如皮质厚度)的比较;维度重复测量如,使用某量表测量同一批研究对象不同生理或心理状态,其不同状态维度之间的比较(如生活质量SF-36量表各个维度之间的比较)。
六、分析小技巧
(一) 球形度检验
重复测量数据的方差分析过程中,需要先考察数据的球形分布特征,当违背了球形假设条件时,需要进行epsilon (ε)校正。epsilon (ε)值越低,说明违反球形假设的程度越大,当epsilon (ε)=1时,完全服从球形假设。当Greenhouse-Geisserepsilonε<0.75时,使用Greenhouse-Geisser方法校正;当Greenhouse-Geisserepsilonε>0.75时,使用Huynh-Feldt方法校正。
(二) 基线数据处理
重复测量数据分析往往会遇到基线数据和后续测量数据的条件不一致的情况,如针对某种治疗方法对镇痛效果的影响,测量了治疗前和治疗后多次时间点的镇痛效果,此时基线数据和后续多个测量数据的条件则不一致。对于这种情况,基线数据一般不应作为重复测量的第一次纳入重复测量方差分析。